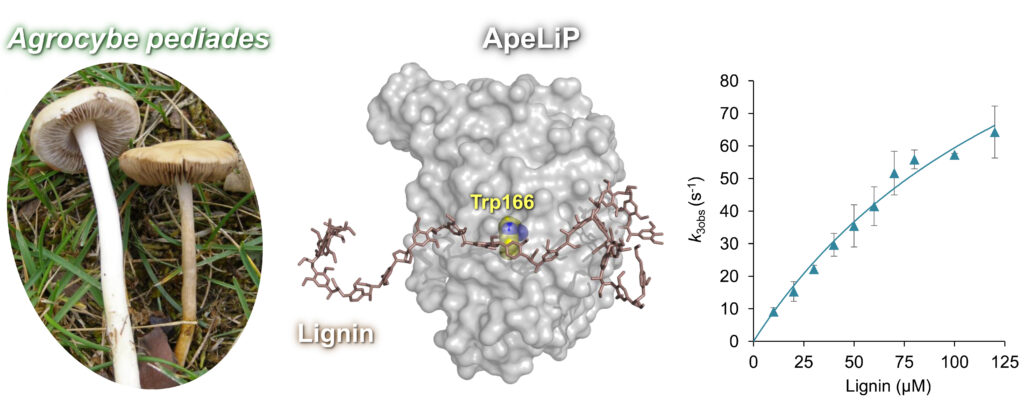
Recreación de la interacción entre el polímero de lignina y el triptófano catalítico de ApeLiP de Agrocybe pediades, y análisis cinético que muestra la capacidad de la enzima para sustraer electrones de la lignina.
Una reciente investigación resultado de la colaboración entre el grupo de «Biomasa vegetal – Aprovechamiento y valorización» del Instituto de Recursos Naturales y Agrobiología de Sevilla (IRNAS) y los grupos de «Biotecnología para la biomasa lignocelulósica» y «Biología estructural de proteínas» del Centro de Investigaciones Biológicas «Margarita Salas» (CIB), publicada en la revista Antioxidants, identifica y caracteriza por primera vez una lignina peroxidasa (responsable de la degradación de la lignina) en un hongo del orden Agaricales, y muestra que es una de las enzimas ligninolíticas más eficientes descritas hasta la fecha.
La degradación de la lignina es esencial para el reciclado de carbono en los ecosistemas terrestres y, a menudo, representa un paso clave para el uso de la biomasa vegetal en la industria. Este proceso se ha estudiado tradicionalmente en el orden Polyporales, donde se incluyen la mayoría de los hongos que degradan la madera. El análisis genómico de estos organismos y la caracterización de sus enzimas han puesto de relieve el papel clave de peroxidasas fúngicas de alto potencial redox (lignina peroxidasas y otras) en el ataque a la lignina. Sin embargo, la degradación de este polímero aromático apenas se ha estudiado en Agaricales, a pesar de ser el orden de basidiomicetos más grande, con alrededor de 13.000 especies descritas, y representar una fuente potencial de nuevas enzimas lignocelulolíticas dada su capacidad para crecer sobre una amplia variedad de materiales lignocelulósicos.
La investigación de decenas de genomas de especies de Agaricales llevada a cabo en el CIB en colaboración con el Joint Genome Institute del Departamento de Energía de EE. UU (DOE-JGI) (https://jgi.doe.gov) ha revelado que genes de lignina peroxidasas aparecieron en este orden fúngico a través de una ruta evolutiva diferente a la que dio lugar a las enzimas de Polyporales. En este trabajo se ha abordado el estudio de la primera de estas peroxidasas descrita en Agaricales, la lignina peroxidasa del hongo Agrocybe pediades (ApeLiP), cuyo genoma ha sido secuenciado recientemente en el marco de un proyecto en colaboración con el DOE-JGI.
El estudio parte de la secuencia del gen de esta enzima y, mediante el empleo de técnicas clásicas y de vanguardia entre las que se incluyen la cristalografía de rayos X, la mutagénesis dirigida, la espectrofotometría de flujo detenido y la resonancia magnética nuclear bidimensional, profundiza en las propiedades estructurales y funcionales de la nueva peroxidasa.
Su estructura cristalográfica, caracterización cinética y estudio de su capacidad ligninolítica revelan el mecanismo catalítico de esta enzima, y muestran que no solo es capaz de oxidar compuestos modelo de lignina, sino también diferentes tipos de lignina real, tanto de angiospermas como de gimnospermas, en un grado similar o incluso superior al de otras peroxidadas ligninolíticas conocidas.
Este trabajo demuestra que no solo los Polyporales degradadores de madera, sino también los hongos Agaricales formadores de setas, tienen enzimas de enorme relevancia tanto para el reciclado del carbono en la naturaleza como para la modificación biotecnológica de la lignina.
Referencia: Agaricales mushroom lignin peroxidase: from structure–function to degradative capabilities. Sánchez-Ruiz MI, Ayuso-Fernández I, Rencoret J, González-Ramírez AM, Linde D, Davó-Siguero I, Romero A, Gutiérrez A, Martínez AT, Ruiz-Dueñas FJ Antioxidants (2021) 10:1446.
https://doi.org/10.3390/antiox10091446
